他の事例をお探しの方は「PC提案事例の検索」をしてみてください。
- 事例No.PC-12218
-
 参考価格:
参考価格:
1,191,300円NGS解析用ワークステーション (2024年11月版)
用途:RNA-seq解析 (Trinity) 、ゲノム解析 (SPAdes、Platanus、Racon、medaka、FALCON、Canu)お客さまからのご相談内容
NGS関連の予算申請のため、解析用PCの必要スペックを相談したい。
用途は、推定280Mbの2倍体生物のゲノムアセンブリで、RNA-seqの解析まで研究を広げる予定がある。予算は100万~120万ほどで、OSはLinux (Ubuntu) を希望。RNA-seq解析ではTrinityの利用を予定している。
ゲノム解析に関しては、アセンブリングに一番スペックが必要になると推測しており、使用ソフトウェアは、ハプロイドのサンプルではSPAdes、Platanus、Racon、medaka、ディプロイドのサンプルではFALCONかCanuを想定。
10Gb程度のデータからアセンブリング~シンテニー解析を行うつもりだが、詳細なソフトまでは決めかねている。
以上の条件に合致するマシンを、予算内で提案して欲しい。
テガラからのご提案
解析用PCの選定においては、計算に十分なメモリ容量の確保を重視するのが一般的な考え方です。そのため、スペック選定の順番とコスト比重は、まずメモリを必要量確保し、残った予算で解析用のCPUやデータ用ストレージを検討する流れとなります。
本件では、10Gb程度のデータから始めるというお話ですので、ご予算120万円の範囲である程度の解析を行うことができる構成をご提案しています。構成のポイントはメモリの拡張性で、「後から+768GBまでは増設できる」点が特徴です。扱うデータ量を段階的に増やす過程で、メモリ不足が発生した場合に増設できる余地のある構成になっています。
注意点として、本事例の構成は解析が可能なメモリ容量を重視しているため、処理速度そのものを重視した構成ではありません。処理速度について条件がある場合は、お気軽にご相談ください。
参考:Trinityのメモリについての表記(Running Trinity · trinityrnaseq/trinityrnaseq Wiki · GitHub)








主な仕様
CPU Intel Xeon W5-3435X 3.10GHz(TB3.0時4.70GHz) 16C/32T メモリ 合計 256GB DDR5 4800 REG ECC 32GB × 8 ストレージ1 1TB SSD M.2 NVMe Gen4 ストレージ2 16TB HDD S-ATA ビデオ NVIDIA T400 4GB (MiniDisplayPort x3) ネットワーク on board (1GbE x1 /10GbE x1) 筐体+電源 タワー型筐体 + 1000W OS Ubuntu 22.04 ■キーワード
・Trinityとは
Trinityは、トランスクリプトームのde novoアセンブリを行うためのソフトウェア。リファレンスゲノムが利用できない生物種や、新規転写産物の発見を目的とする場合に有効。RNA-Seqのリード(短い塩基配列)を使って、元のmRNA配列を復元することができる。
・SPAdesとは
SPAdesは、ゲノム配列のde novoアセンブリを行うためのソフトウェア。次世代シーケンサー (NGS) データを用いてゲノム配列を再構築するためのアセンブラで、特にバクテリアゲノムのアセンブリに適している。シングルセルシーケンシング (SCS) データにも対応している。
参考:GitHub – ablab/spades: SPAdes Genome Assembler ※外部サイトに飛びます
・Platanusとは
Platanusは、ゲノム配列のde novoアセンブリを行うためのソフトウェア。特に高度に異質性のあるゲノムのアセンブリに適したツールで、次世代シーケンサーのショートリードデータを使用して、高精度なゲノム配列を再構築することができる。
参考:GitHub – rkajitani/Platanus_B: De novo genome assembler for bacterial genomes ※外部サイトに飛びます
・Raconとは
Raconは、ロングリードシーケンスデータを用いたde novoゲノムアセンブリにおいて、高速かつ正確なコンセンサス配列を生成するためのツール。エラー率の高いロングリードから高品質なコンセンサス配列を迅速に生成することを目的としており、特にPacBioやOxford Nanopore Technologiesのデータに適している。
・medakaとは
medakaは、次世代シーケンシングデータの解析、特にDNAの変異検出に使用されるツール。主にOxford Nanopore Technologies (ONT)のロングリードシーケンスデータを対象としたポリッシングと変異検出のためのツールでで、特に、de novoアセンブリの後処理や、既知のリファレンスゲノムに対する変異コールに適している。
参考:GitHub – nanoporetech/medaka: Sequence correction provided by ONT Research ※外部サイトに飛びます
・FALCONとは
FALCONは、PacBioのロングリードシーケンスデータを用いたde novoゲノムアセンブリを行うためのソフトウェア。Pacific Biosciences (PacBio) 社が開発したde novoゲノムアセンブラーで、大規模で複雑なゲノムのアセンブリに適している。
・canuとは
Canuは、ロングリードシーケンスデータを用いたde novoゲノムアセンブリを行うためのソフトウェア。PacBioやOxford Nanopore Technologiesのロングリードデータに特化したde novoアセンブラーで、大規模で複雑なゲノムのアセンブリに適している。
事例追加日:2024/11/06
ご注文の流れ
 |
お問い合わせフォームよりご相談内容をお書き添えの上、 お問い合わせください。 (お電話でもご相談を承っております) |
 |
弊社より24時間以内にメールにてご連絡します。 |
 |
必要に応じてメールにて打ち合わせさせていただいた上で、 メール添付にてお見積書をお送りします。 |
 |
お見積もり内容にご納得いただけましたら、メールにてご注文ください。 ご注文確定後、必要な部材を手配し PCを組み立てます。 (掛売りの場合、最初に新規取引票のご記入をお願いしております) |
 |
動作チェックなどを行い、納期が確定いたしましたらご連絡いたします。 (納期は仕様や製造ラインの状況により異なります) |
 |
お客様のお手元にお届けいたします (ヤマト運輸/西濃運輸) |
お支払い方法
お支払い方法は、お見積もりメール・お見積書でもご案内しています。
| 法人掛売りのお客様 |
| 原則として、月末締、翌月末日払いの後払いとなります。 |
| 学校、公共機関、独立行政法人のお客様 |
| 納入と同時に書類三点セット(見積書、納品書、請求書)をお送りしますのでご請求金額を弊社銀行口座へ期日までにお振込み願います。 先に書面での正式見積書(社印、代表者印付)が必要な場合はお知らせください。 |
| 企業のお客様 |
| 納品時に、代表者印つきの正式書類(納品書、請求書)を添付いたします。 ご検収後、請求金額を弊社銀行口座へお支払い期日までにお振込み願います。 |
| 銀行振込(先振込み)のお客様 |
| ご注文のご連絡をいただいた後、お振込みを確認した時点で注文の確定とさせていただきます。 |
修理のご依頼・サポートについて
弊社製PCの保証内容は、お見積もりメールでもご案内しています。
■お問合せ先
テガラの取り扱い製品に関する総合サポート受付のWEBサイトをご用意しております。
テガラ株式会社 サポートサイト
※お問い合わせの際には、「ご購入前」と「ご購入後」で受付フォームが分かれておりますので、ご注意ください。
| メール | support@tegara.com |
| 電話 | 053-543-6688 |
■テグシスのサポートについて
保証期間内の修理について
保証期間内におけるハードウェアの故障や不具合につきましては、無償で修理いたします。
ただし、お客様による破損や、ソフトウェアに起因するトラブルなど保証規定にて定める項目に該当する場合は保証対象外となります。
保証期間経過後も、PCをお預かりしての初期診断は無料で実施しております。
無料メール相談
PCの運用やトラブルにつきまして、メールでのご相談を承ります。経験・知識の豊富な技術コンサルタントが無料でアドバイスいたします。
※調査や検証が必要な場合はお答えできなかったり、有償対応となることがあります
オプション保証サービス
| 「あんしん+」 もしもの時の延長保証サービス |
|
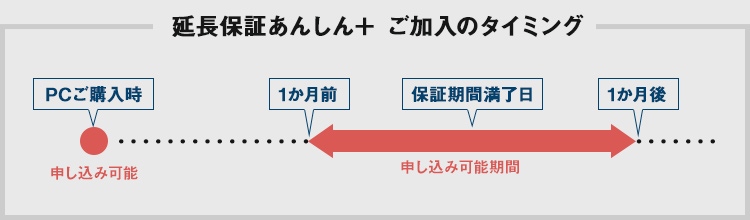
PCのご購入時にトータル5年までの延長保証をご選択いただけます。また、ご購入後にも延長保証を申し込むことができます。
|
| HDD返却不要サービス |
|
保証期間内にPCのHDD(SSD)が故障した場合、通常、新品のHDDとの交換対応となり、故障したHDDはご返却いたしません。 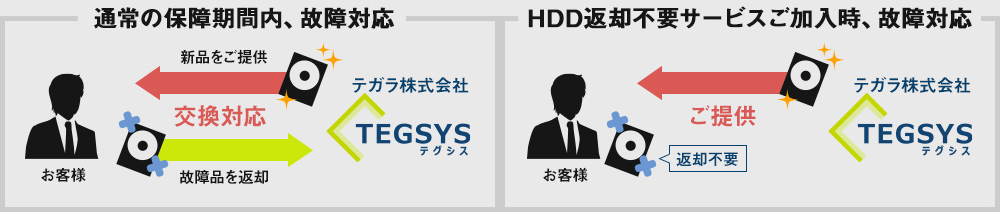
|
| オンサイト保守サポート | |
|
故障発生時、必要に応じエンジニアスタッフが現地へ訪問し、保守対応を行うサービスです。
|
「お客様だけのオーダーメイドPC」を製作しています。
用途に応じた細かなアドバイスや迅速な対応がテガラの強みです。